问题描述
定义了一组药物R = r1,r2,…,rm和一组疾病D = d1,d2,…,dn,其中m和n是R中的药物数量和D中的疾病,分别。对于R,我们基于不同的药物相关数据集将一组药物之间的相似性邻接矩阵定义为SR,其中SR = sr1,sr2,…,srk和k是药物相关数据集的数量。每个药物相似性邻接矩阵的维为m×m; 其中m是k个药物相关数据集中不同药物的数量,而sra(ru,rw)表示基于药物相关数据集a的ru和rw这对药物相似。
类似地,让疾病之间的相似性邻接矩阵D集基于不同的疾病相关数据集定义为SD,其中SD = sd1,sd2,…,sdl,其中l是疾病相关数据集的数量。每个疾病相似度邻接矩阵的维数为n×n;其中n是与l个疾病相关的数据集中不同疾病的数量。 sdb(dx,dz)的值指示基于疾病相关数据集b的一对疾病dx和dz相似。SD相似性邻接矩阵中的所有值都在[0,1]范围内,其中0表示绝对不同,而1表示一对疾病之间的完全相似。
接下来,将药物组R和疾病组D之间的相互作用定义为m×n的二元矩阵Y,如果药物ri与疾病dj相互作用,则yij = 1,如果未知则yij = 0药物ri与疾病相互作用的证据dj。给定矩阵Y以及两组矩阵SR和SD,目标是预测Y中新型的药物-疾病相互作用。
SNF‑NN方法的说明
SNF-NN方法是一种新颖的方法,可通过利用与药物相关的相似性信息,与疾病相关的相似性信息和已知的药物-疾病相互作用来深入了解药物疾病的相互作用。 SNF-NN集成了相似性度量,相似性选择,相似性网络融合(SNF)和神经网络(NN),并执行了非线性分析,从而提高了药物-疾病相互作用的预测准确性。
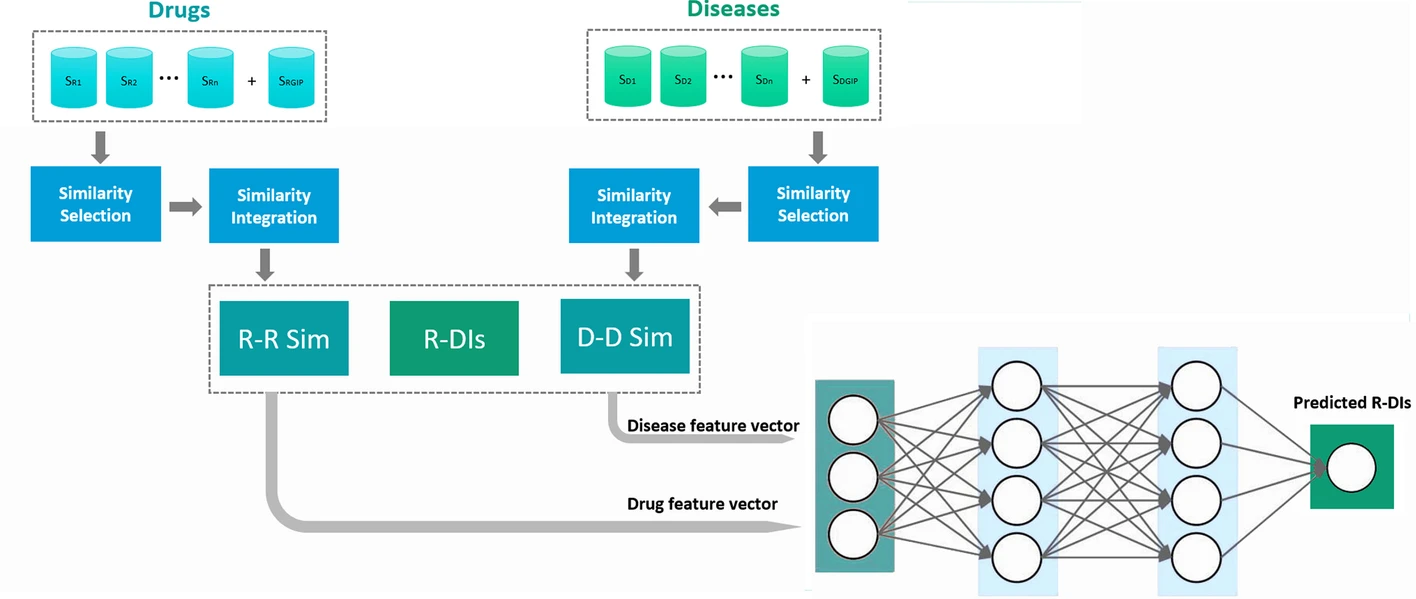
SNF-NN方法由四个步骤组成。在第一步中,它从各种来源获取与药物相关的信息(例如,药物靶蛋白、药物化学结构、药物副作用)、与疾病相关的信息(例如,疾病-基因关联、疾病-miRNA关联、疾病表型)以及已知的药物-疾病相互作用信息。接下来,它利用每种药物相关或疾病相关信息类型的最多文献认可的相似性度量来计算该特定药物相关或疾病相关信息类型的成对药物或疾病相似性。此外,它还根据已知的药物-疾病相互作用计算药物对和疾病对的高斯相互作用轮廓(GIP)相似度。在第二步中,它使用启发式过程来选择最有洞察力且冗余较少的药物和疾病相似类型的子集。第三步,利用非线性相似网络融合方法,对筛选出的药物和疾病相似类型进行融合。在第四步中,它首先对融合的药物和疾病相似性信息进行笛卡尔乘积,将每个药物-疾病对的特征向量连接起来。最后,它将串联的特征向量和已知的药物-疾病相互作用馈送到多层神经网络,以预测输入的药物-疾病对之间的新的相互作用。SNF-NN方法的总体工作流程如图1所示。
计算相似性度量
为每个与药物相关和与疾病相关的数据集计算成对相似度,以便量化每个药物对或疾病对之间的共同特征。对于每种与药物和疾病相关的信息类型,采用了最多文献认可的相似性度量。药物和疾病成对相似度值在一对药物或疾病之间的范围[0,1]内,其中0表示最小相似,1表示最相似。此外,假定在已知的药物-疾病相互作用中与疾病相互作用的一对药物也会表现出与新疾病类似的行为。同样,假定在已知的药物-疾病相互作用中,一对与药物相互作用的疾病在被新药物治愈后,将以同样的方式表现出来。因此,我们利用药物-疾病相互作用信息来计算高斯相互作用谱核[40],也称为径向基函数(RBF)核,作为药物对或疾病对之间的谱相似度。
药物-疾病相互作用数据集中的每种药物r用数据集f®中的疾病的二值特征向量表示,其中疾病相互作用的存在或不存在分别用0或1编码。
类似地,药物-疾病相互作用数据集中的每种疾病d都用数据集g(d)中药物的二值特征向量表示,其中药物相互作用的存在或不存在分别用0或1编码。
通过分别除以每种药物的疾病相互作用和每种疾病的药物相互作用的平均数量,对药物和疾病的两两相似度值进行归一化。
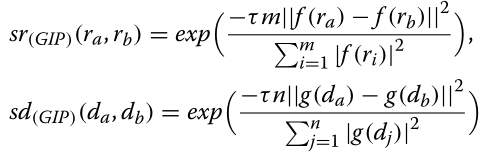
其中参数τ控制核带宽,m和n为药物-疾病相互作用数据集中药物和疾病的总数量,|f(ri)|为药物ri的疾病相互作用数量,|g(dj)|为疾病dj的药物相互作用数量。这里,正如van Laarhoven等人所指出的,τ被简单地设置为1。
相似性的选择
SNF-NN适用于任何一组与药物相关和与疾病相关的相似类型。然而,当试图整合这些不同的相似类型时,这些药物相关和疾病相关相似类型的质量、丰富度和相关性是至关重要的。数据不一致和冗余可能会导致集成的药物相关相似矩阵产生噪声。因此,我们使用了Olayan等人提出的一种有效的方法[41],选择信息最丰富、最有洞察力、冗余最少的药物相似性度量子集组合。该启发式相似性选择过程包括四个步骤,并在[42]中进行了解释。
【42】Jarada TN, Rokne JG, Alhajj R. SNF-CVAE: computational method to predict drug-disease interactions using similarity network fusion and collective variational autoencoder. Knowl Based Syst. 2020;212:106585.
fig2. 选取信息最丰富且冗余度最小的药物相似矩阵子集的过程流程图。
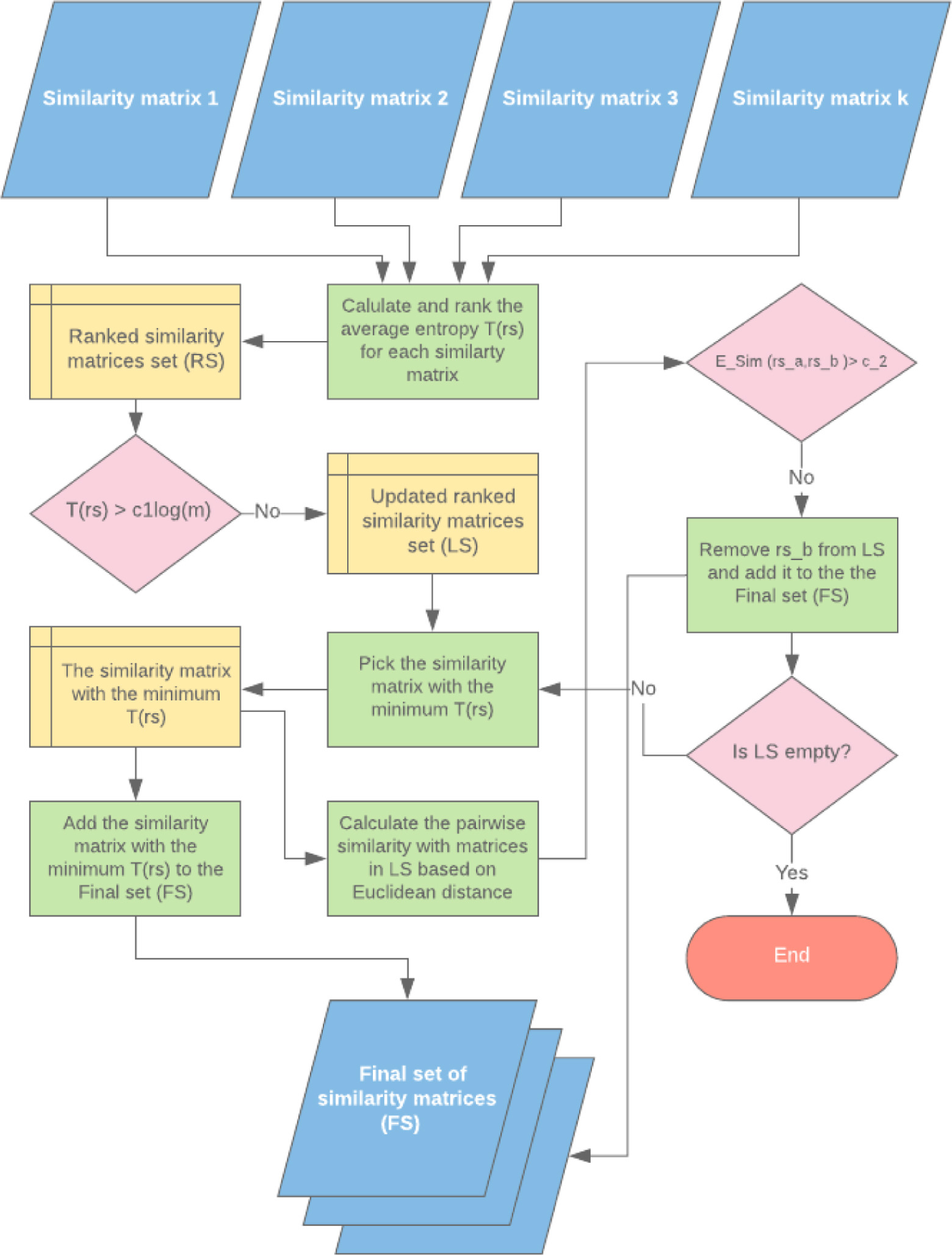
1. 计算每个相似矩阵的平均熵
一个与药物相关的相似类型的平均熵表明了特定药物相关数据集中携带信息的程度。设rs为大小为m × m的药物相关相似矩阵,其中m为药物个数,rij为药物ri与rj的相似值。平均熵T(rs)计算为:
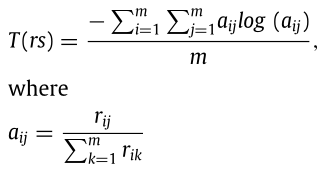
2,消除信息较少的相似矩阵
相似矩阵的平均熵值反映了矩阵中信息的随机性水平和信息的信息量。因此,平均熵值越大,相似矩阵的信息量就越小。对药物相关相似矩阵按照其平均熵值从小到大排序,并剔除平均熵值大于阈值的相似矩阵。
阈值定义为c1log(m),其中c1控制要选择的熵值水平,m为相似矩阵中药物的个数,log(m)表示可能的最大熵值。
通过检查不同的c1值,评估本文方法的整体性能,在c1 = 0.7时获得最佳结果。
3,计算所选相似矩阵之间的成对相似度
利用欧几里得距离计算药物相关相似矩阵之间的成对相似度,以确定任意一对相似矩阵之间可能存在的数据冗余和信息重叠。给定一对大小为m × m的药物相关相似矩阵rsa和rsb,定义一对药物相关矩阵Esim(rsa, rsb)的两两相似度为:


4,消除冗余相似矩阵
通过对2.4节中选择的相似矩阵进行迭代,得到最后一组信息最丰富且冗余度最小的相似矩阵。
假设LS = ls1, ls2,…, lsz为步骤2中选取的相似矩阵的排序集,FS =为最终选取的相似矩阵集。
在第一次迭代中,将具有最小平均熵值的相似矩阵,lsa添加到FS中,当 lsb 满足Esim(rsa, rsb) > c2时,将相似矩阵 lsb 从 LS 中移除,其中c2为常数。
这个过程将在更新的LS集上迭代,直到它变为空为止。常数c2是一个阈值,它控制任意一对选定的相似矩阵之间允许的相似/冗余程度。通过检查不同的c2值,并对所提方法的整体性能进行评价,c2 = 0.6时获得最佳结果。
最终,所选相似矩阵的最终集合FS包含了相似矩阵输入集合的高信息量和少冗余相似矩阵的子集。
相似网络融合
这一步骤的目标是将前一步骤中信息丰富且冗余较少的相似矩阵集成到一个全面的药物相似矩阵中,该相似矩阵从整个相似矩阵集合中获取共享信息和任何互补知识。因此,在给定一组药物相似矩阵的情况下,我们利用Wang等人[43]提出的相似网络融合方法构造一个融合的相似矩阵。SNF方法利用基于消息传递理论的迭代非线性过程,将给定的药物相似矩阵集合合并为一个综合矩阵。SNF迭代应用k近邻(KNN)算法,用其他药物相似矩阵的信息更新每个药物相似矩阵,直到得到一个尽可能好的表示初始药物相似矩阵集的药物相似矩阵。
【43】Wang B, Mezlini AM, Demir F, Fiume M, Tu Z, Brudno M, Haibe-Kains B, Goldenberg A. Similarity network fusion for aggregating data types on a genomic scale. Nat Methods. 2014;11(3):333.
给定上一步中选择的药物相关和疾病相关相似矩阵的子集,这一步的目标是将这些选择的相似矩阵整合成两个药物和疾病的综合相似矩阵。因此,给定一组药物和疾病的多个相似矩阵,应用迭代非线性相似网络融合过程,分别构建两个融合的药物-药物和疾病相似矩阵。这两个融合的相似矩阵分别从药物和疾病的相似矩阵中获取共享信息和任何互补知识。
神经网络模型
神经网络结构定义对神经网络模型的预测性能有重要影响。前馈多层感知器网络结构作为一种有效的神经网络结构,近年来受到计算生物学领域的广泛关注。这样的神经网络中的信息只能在一个方向上从输入层向前移动,通过隐藏层到达输出层;因此,网络中没有循环或环路。输入层包含神经网络的初始数据;隐藏层是进行所有计算的中间层,输出层产生给定数据输入的结果。神经网络的每一层都由许多计算神经元或单元组成。一个神经元与下一层的一组神经元相连。每个神经元都有一个权值,它可以定义为神经元对下一层神经元的影响。每个神经元的输入值乘以神经元的权重,得到传递到下一层的神经元的输出值。神经网络模型的性能和学习速度高度依赖于隐含层的数量,以及每个隐含层中神经元的数量。为了进一步提高深度学习预测药物-疾病相互作用的准确性,本文引入了一种全连接的前馈多层感知器网络模型。
为了获得最佳的整体精度,需要对深度神经网络模型的各种超参数进行繁琐的调整。具体来说,需要指定隐含层的数量,每层神经元的数量,每层背后应用的激活函数类型,以及每层[44]的学习退出率。此外,其他超参数范围从α, β1, β2,和 ǫ 为亚当优化算法[45]的权重和偏差参数[46], epoch的数量,和批大小可能需要检查。
值得一提的是,每个超参数的重要性取决于训练数据,有些超参数可能比另一些更重要。例如,玩弄隐藏层的数量、每层中的神经元数量或学习速率有时会产生巨大的不同。
【47】Cawley GC, Talbot NL. On over-fitting in model selection and subsequent selection bias in performance evaluation. J Mach Learn Res. 2010;11(Jul):2079–107.
为了确定神经网络的结构,使用嵌套交叉验证[47]系统地组织超参数的调优过程,使其在收敛到合适的超参数设置时更加高效。在不使用嵌套交叉验证的情况下应用超参数调优过程将导致模型过拟合,因为在调优和评估模型性能时使用的是相同的数据。嵌套交叉验证中的超参数调整如下:
(1)模型超参数设置为特定值。
(2)将输入数据集分成三个部分。
(3)使用当前超参数值和两份数据集对模型进行训练。
(4)使用所选择的超参数值和剩余的数据集(测试集)对模型进行测试。
(5)重复步骤3和4,直到每份数据集被认为是测试集。
(6)记录模型性能结果和超参数值组合。
(7)对超参数值的所有组合执行步骤1至6。
(8)选择综合性能最好的超参数值组合。
以下超参数和关联值用于确定神经网络模型体系结构:
·隐藏层数量:{1,2,3,4,5}
·每个隐藏层神经元数量:{100,200,300,400,500}
·激活函数:
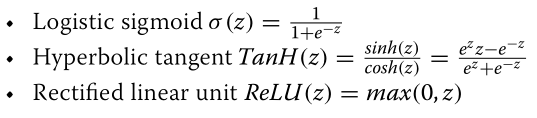
• Dropout rate: {0.3, 0.35, 0.4., 0.45, 0.5}
采用4个隐层、300个神经元组成的深度神经网络模型取得了最好的效果,每层的Dropout率为0.35。将整流线性单位激活函数应用于所有隐藏层的所有神经元。由于预测药物-疾病相互作用是一个二值分类问题,因此在输出层应用logistic sigmoid激活函数,并使用二值交叉熵损失函数计算损失值。
使用He初始化[48]初始化权值和偏差参数,并用Adam优化算法更新这些参数时,深度神经网络模型的效果最好。
根据[45]的建议,Adam的hyperparameters设置如下:
• The learning rate (α) : 1E−3.
• The exponential decay rate for the first-moment estimates (β1) : 0.9
• The exponential decay rate for the second-moment estimates (β2) : 0.999
• The small constant for numerical stability (ǫ) : 1E−7
最后,向模型输入批量为100的批量输入。对于不同的数据集,epoch的数量被设置为100。
所有训练过的深度神经网络模型及其超参数设置和嵌套交叉验证结果的性能都在附加文件1中给出。
结果的差异
Spec度量的定义是为了评估机器学习方法在预测负关系(即无rdi)方面的性能,而SNF-NN则侧重于预测正关系(即新rdi)。
因此,就规格评估指标而言,SNF-NN没有最高的性能并不重要。
最后,值得强调的是,F1度量被定义为Prec和Rec的调和平均值;
因此,当SNF-NN具有最好的F1值时,其对Prec和Rec的性能并不是最高的,这并不奇怪。
实验过程
在此,描述了验证SNF-NN鲁棒性和预测性能的系统评价准则。
首先,进行10倍交叉验证,并将SND、Cdataset和LRSSL数据集的每个金标准分为训练集和测试集,以防止任何对模型性能的过于乐观的评估。
在分层的10倍交叉验证过程中,已知药物-疾病相互作用(即,正相关)与匹配数量的未知药物-疾病相互作用(即,负相关)被随机分成10组。
每一组都有相同数量的正负关系。
在每个交叉验证试验,九集被反过来作为训练集,而其余组代表了10倍交叉验证测试集。与不同的随机种子值,重复了五次,平均性能计算,以避免任何高方差和偏置交叉验证评估。